Komisja do spraw Specjalizacji Lekarzy Weterynarii przy Państwowym Instytucie Weterynaryjnym –…
Zapraszamy do udziału w szkoleniu pt. „Wybrane aspekty związane z pracą lekarza weterynarii”, które…
Z głębokim smutkiem i niedowierzaniem przyjęliśmy wiadomość o śmierci Dr. n. wet. Artura…
W dniach 17-18 lutego br. przedstawiciele Instytutu: prof. dr hab. Mirosław P. Polak, zastępca…
Państwowy Instytut Weterynaryjny – Państwowy Instytut Badawczy rozpoczął proces wdrażania zasad…
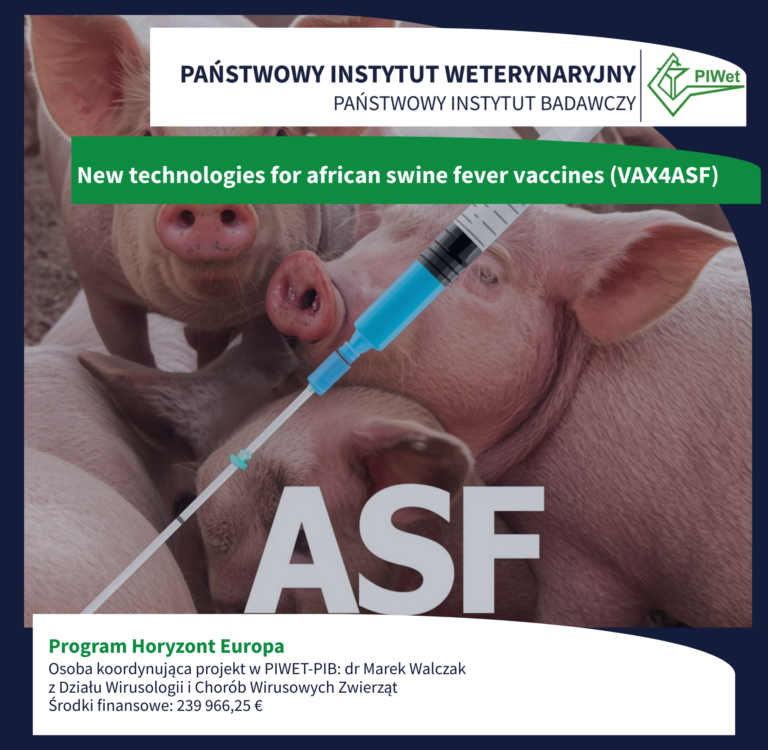
Państwowy Instytut Weterynaryjny – Państwowy Instytut Badawczy we współpracy z 16 partnerami z 10…
W czerwcu ubiegłego roku, podczas Konferencji w Korytnicy, odbył się benefis dr. n. wet. Jana…
4 lutego br. odbyło się inauguracyjne posiedzenie Rady Naukowej Państwowego Instytutu…
Informujemy, że najnowsze dane dotyczące sytuacji epizootycznej w Polsce oraz aktualizację działań…
Z głębokim żalem zawiadamiamy o śmierci dr. Jerzego Krasuckiego (ur. 1949) wieloletniego pracownika…
Zdrowych, spokojnych i pełnych rodzinnego ciepłaŚwiąt Bożego Narodzenia,chwil wytchnienia, radości i…
Uprzejmie informujemy, że dni 2 i 5 stycznia będą w Państwowym Instytucie Weterynaryjnym –…
Projekt Krajowego Laboratorium Referencyjnego ds. włośnicy Działu Parazytologii i Chorób…
Świadome podejście do bezpieczeństwa zaczyna się od ludzi, dlatego PIWet-PIB konsekwentnie podnosi…
W dniu 12 grudnia br. w siedzibie Narodowego Instytutu Kultury i Dziedzictwa Wsi w Warszawie odbyła…
W dniach 9–10 grudnia 2025 r. odbyła się kursokonferencja „Realizacja badań chemicznych w urzędowej…
W dniu 6 grudnia 2025 r. odbyło się uroczyste wręczenie dyplomów specjalisty dla lekarzy…
W dniu 8 grudnia br. w siedzibie Głównego Inspektoratu Sanitarnego w Warszawie odbyła się…
Głębokim smutkiem napełniła nas wiadomość o śmierci w dniu 4 grudnia br. Prof. dr. hab. dr. h.c…
Z przyjemnością informujemy, że projekt pt. „PFAS z kranu na stół: Czy skażona woda zwiększa…
W dniu 26 listopada br. dyrektor PIWet-PIB prof. dr hab. Stanisław Winiarczyk podpisał umowę o…
26 listopada 2025 r. w PIWet-PIB odbyło się uroczyste otwarcie zmodernizowanej zwierzętarni…
W dniach 19–20 listopada 2025 r. w Brukseli odbyło się doroczne spotkanie sieci laboratoriów OMCL…
W dniu 23 października 2025 r. w Państwowym Instytucie Weterynaryjnym – Państwowym Instytucie…
Idąc naprzeciw oczekiwaniom naszych Klientów, Państwowy Instytut Weterynaryjny – PIB w Puławach…
W dniu 18 października 2025 roku na terenie Państwowego Instytutu Weterynaryjnego – Państwowego…
21 października 2025 r. minęło 100 lat od urodzin prof. dr hab. Jadwigi Juśko-Grundboeck…
W dniach 14-15 października 2025 w siedzibie Instytutu Zootechniki-Państwowego Instytutu Badawczego…
W dniach 13–14 października 2025 r. w naszym Instytucie odbyło się szkolenie pt. „Problemy zdrowotne…
W dniach 13–14 października 2025 r. w Państwowym Instytucie Weterynaryjnym – Państwowym Instytucie…
28 września 2025 r. Państwowy Instytut Weterynaryjny – Państwowy Instytut Badawczy w Puławach wziął…
Poszukujemy osoby z tytułem magistra biologii, biotechnologii, weterynarii, mikrobiologii lub…
Weź udział w V ogólnopolskim konkursie filmowym „Świat się kręci wokół wsi” Zapraszamy do udziału w…
Za nami IV Kongres Nauk Rolniczych (10-11 września 2025), organizowany wspólnie przezInstytut Uprawy…
W dniach 5–7 września 2025 r. Państwowy Instytut Weterynaryjny – PIB uczestniczył w XXXIV Krajowej…
Szanowni Państwo, Zapraszamy na 26. Kongres IOM w Krakowie! Z radością informujemy, że w dniach 6–10…
W dniu 22 sierpnia 2025 r. dr Małgorzata Warenik-Bany z PIWet– PIB pełniła rolę recenzentki podczas…
28 sierpnia 2025 r., po 28 latach pracy, swoją zawodową drogę w Państwowym Instytucie Weterynaryjnym…
W dniach 14–15 sierpnia 2025 r. w Reykjaviku (Islandia) odbyła się 11. Konferencja…
Szanowni Państwo, Informujemy iż, zakończony został Projekt OrgSafety „Wprowadzenie innowacyjnej…
W dniu 7 sierpnia 2025 r. przedstawiciele Dyrekcji Instytutu w osobach prof. Stanisława Winiarczyka…
W dniach 6–8 sierpnia 2025 r. Państwowy Instytut Weterynaryjny – Państwowy Instytut Badawczy w…
Z końcem lipca 2025 roku na zasłużoną emeryturę odeszły nasze Koleżanki: mgr Grażyna Krzyżostaniak…
Mgr Małgorzata Samorek-Pieróg reprezentowała PIWet-PIB podczas Europejskiej Szkoły Letniej EUP…
24 lipca br. odbyły się w PIWet-PIB wybory do Rady Naukowej (RN) Instytutu na lata 2025-2029 (XII…
Z wielką dumą informujemy, że Profesor Jacek Osek, kierownik Działu Badań Mikrobiologicznych…
W dniach 14–17 lipca 2025 r. dr hab. Jolanta Grażyna Rola, profesor instytutu, wzięła udział w…
WAŻNE: W okresie od 22.07.2025 – do 22.08.2025 badania w kierunku określenia poziomu…
PIWet-PIB z kolejnym krokiem ku innowacji Z przyjemnością informujemy, że Państwowy Instytut…
PIWet-PIB z kolejnym krokiem ku innowacji Z przyjemnością informujemy, że Państwowy Instytut…